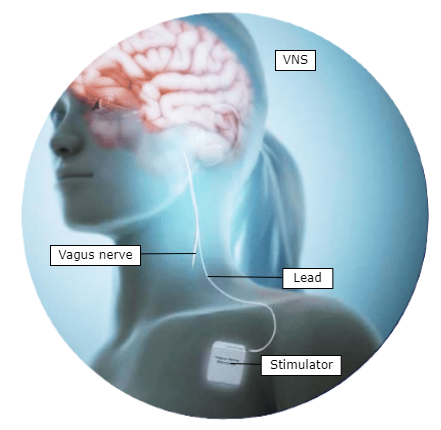
La stimulation du nerf vague (VNS) représente une innovation prometteuse dans le traitement de maladies chroniques, étant déjà utilisée chez un grand nombre de patients souffrant d’épilepsie, de dépression, d’obésité, d’arythmies cardiaques, entre autres. Dans ses applications cliniques actuelles, les patients reçoivent une dose statique de stimulation électrique appliquée au nerf, laquelle doit être ajustée individuellement par un professionnel de santé lors de visites en clinique. Des études préliminaires suggèrent que les thérapies en boucle fermée, réactives au patient, dans lesquelles un dispositif ajuste automatiquement les schémas de stimulation pour une intervention en temps réel et personnalisée, pourraient améliorer considérablement les résultats cliniques.
Le développement d’un système de neuromodulation en boucle fermée est complexe, notamment en raison des grandes quantités de données échangées entre le cerveau et les organes, qui doivent être décodées et exploitées en boucle fermée pour déterminer les stratégies de stimulation optimales. Lors de l’enregistrement d’un signal nerveux, l’un des défis majeurs est d’identifier le type de fibre nerveuse d’où provient le signal ; cette information est cruciale pour comprendre quel organe ou mécanisme physiologique est affecté.
Dans ce travail, nous utilisons l’apprentissage automatique pour identifier automatiquement quelles fibres nerveuses sont recrutées dans le nerf vague suite à une stimulation électrique. Le protocole que nous présentons constitue une preuve de concept en vue d’un processus où une acquisition massive de données pourrait alimenter des thérapies de stimulation du nerf vague en temps réel. Dans le cadre de ce protocole, nous proposons également un outil d’annotation permettant de visualiser et d’annoter les réponses segmentées, afin de créer un ensemble de données plus riche.
Contexte sur la segmentation des eCAP
VNS (Stimulation du nerf vague) : La VNS est une technique thérapeutique qui repose sur la délivrance d’impulsions électriques au nerf vague. Ce processus implique généralement l’implantation chirurgicale d’un petit dispositif, similaire à un pacemaker, connecté au nerf vague. En modulant l’activité de ce nerf, les cliniciens peuvent influencer les voies neuronales impliquées dans la régulation de l’humeur, le contrôle des crises, la modulation de l’inflammation, le fonctionnement du système nerveux autonome, ainsi que d’autres fonctions physiologiques.
eCAP (Potentiels d'action composés évoqués) : Les fibres nerveuses s’activent sous forme de potentiels d’action, tout comme les neurones du cerveau. La somme des potentiels d’action provenant d’axones distincts est appelée potentiel d’action composé (CAP). Ainsi, la réponse neuronale faisant suite à la stimulation du nerf vague est caractérisée par des CAP évoqués (eCAP), qui sont au cœur de l’efficacité de la VNS.
Dans le cadre de la thérapie de neuromodulation, pour produire l’effet thérapeutique souhaité lors de la stimulation, différents eCAP doivent être déclenchés afin de permettre le contrôle de l’activation des différents types de fibres nerveuses, dans le but d’observer leurs effets sur la physiologie. Voir Berthon (2023) [1].
La segmentation automatique des eCAP consiste à diviser la réponse neuronale enregistrée (sous forme de série temporelle) en segments distincts, chacun correspondant à un type de fibre spécifique activé par le stimulus. En apprentissage automatique, ce problème peut être formulé comme un problème de segmentation unidimensionnelle de séries temporelles. Nous concevons nos modèles de manière à intégrer les paramètres expérimentaux influents tels que l’intensité du courant (mA), la largeur d’impulsion de stimulation (μs) et la polarité de stimulation (anodique/cathodique).
Jeu de données
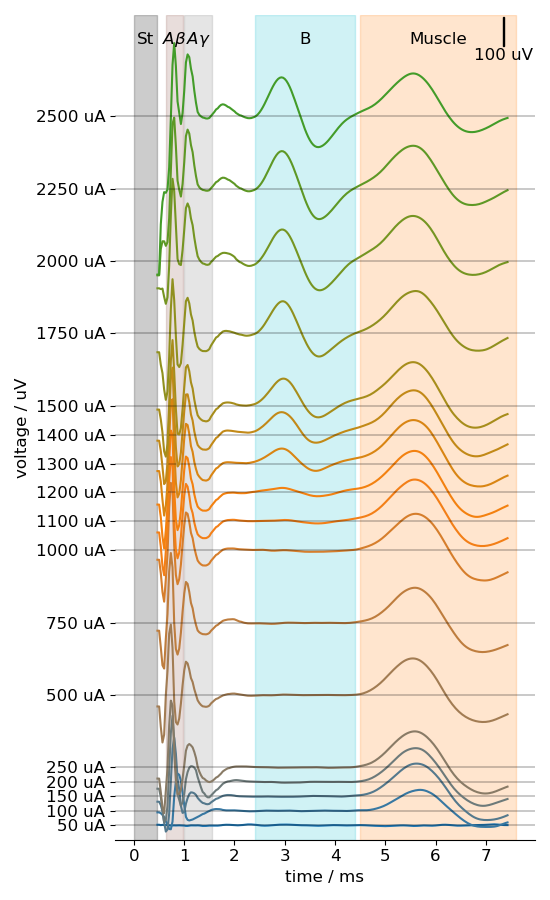
Chaque neurogramme est une série temporelle composée de 290 points de données, représentant l’activation nerveuse en microvolts (μV) dans un modèle animal. Les bornes annotées manuellement marquant le début et la fin de l’activation des différentes fibres nerveuses ont été converties en un masque d’étiquettes continues. Ainsi, chaque point de données est associé à une étiquette entière représentant le type de fibre auquel il appartient. Les points ne correspondant à aucun des quatre types de fibres d’intérêt ont été codés avec une étiquette générique appelée « autre ».
Le jeu de données comprend 129 768 échantillons.
Les données utilisées dans les expériences actuelles ont été annotées manuellement par des experts, qui ont observé les schémas d’activation de différentes fibres à l’aide d’un graphe empilé similaire à la Figure 2.
Méthodes
Référence de base
La méthode de référence simple consiste à moyenner les limites de début et de fin annotées manuellement pour chaque type de fibre, en se basant sur les sujets de l’ensemble d’entraînement. Tous les échantillons de test reçoivent alors les mêmes bornes pour chaque type de fibre. Cette approche établit une référence raisonnable basée sur les données, atteignant un score F1 macro moyen de 0,37.
Approches proposées pour la segmentation des eCAP
Aucun travail antérieur n’existe à ce jour sur la segmentation automatisée des eCAP à l’aide de l’apprentissage automatique. Nous avons donc développé et comparé trois modèles différents. En nous inspirant des modèles de segmentation d’images basés sur des architectures encodeur-décodeur, nous avons formulé notre problème d’apprentissage de manière similaire, où le modèle apprend à convertir une séquence d’entrée en une séquence d’étiquettes prédites pour chaque point.
Les modèles basés sur LSTM (Long Short-Term Memory) ont démontré leur efficacité dans la prédiction de séquences et d’autres tâches liées aux séries temporelles. Nous avons commencé par une architecture BiLSTM (bidirectionnelle), inspirée des travaux existants en segmentation de signaux ECG.
Ensuite, nous avons exploré deux architectures encodeur-décodeur de type séquence-à-séquence :
- LSTM-ED (Encodeur-Décodeur basé sur LSTM)
- Conv-ED (Encodeur-Décodeur basé sur des convolutions 1D)
L’intégration de couches d’attention multi-têtes (multi-headed attention) permet au modèle de comparer simultanément les différentes fibres présentes dans la séquence d’entrée, tout en facilitant la combinaison des caractéristiques numériques.
Normalisation et partition des données
En raison de la variabilité inter-individuelle, nous calculons les paramètres de normalisation pour chaque sujet individuellement, plutôt que sur l’ensemble d’entraînement global. De manière similaire, pour reproduire une situation d’usage réel, où le modèle doit inférer sur un sujet totalement inconnu, nous construisons les partitions de validation de manière à ce que les sujets de validation et de test soient indépendants.
Résultats
Les Tableaux 1 et 2 présentent deux niveaux de résultats résumés :
- Le Tableau 1 affiche les moyennes pondérées des scores F1 ainsi que les scores F1 macro moyens pour tous les types de fibres.
- Le Tableau 2 indique la moyenne des scores F1 pour les quatre types de fibres nerveuses.
L’étiquette « autre » a été exclue du calcul de ces scores, car elle ne correspond à aucun type de fibre.
D’après le Tableau 2, on observe que les fibres A-bêta et A-gamma sont plus faciles à prédire que les autres. Cela s’explique par la régularité de leur activation, tant en position qu’en forme, dans la majorité des échantillons. À l’inverse, le manque de cohérence dans l’activation des fibres B selon les sujets fait chuter significativement la moyenne de leurs performances de prédiction.
Globalement, le modèle BiLSTM avec attention (BiLSTMs+Attn) obtient les meilleurs résultats, dépassant légèrement la référence de base. Le modèle Conv-ED ne donne pas de bons résultats, tandis que LSTM-ED n’apporte pas une amélioration suffisante par rapport à la baseline.
| Model | Macro F1 Score | Weighted Average F1 |
| Baseline | 0.37 | 0.40 |
| BiLSTM+Attn | 0.485 | 0.498 |
| LSTM-ED | 0.405 | 0.485 |
| Conv-ED | 0.331 | 0.391 |
Tableau 1: Scores F1 macro et F1 macro pondéré par modèle
| Model | A-beta | A-gamma | B | A-delta |
| Baseline | 0.58 | 0.53 | 0.36 | 0.03 |
| BiLSTM+Attn | 0.635 | 0.590 | 0.442 | 0.123 |
| LSTM-ED | 0.601 | 0.541 | 0.433 | 0.032 |
| Conv-ED | 0.520 | 0.501 | 0.297 | 0.023 |
Tableau 2: Scores F1 moyens par modèle et par type de fibre
| Class | Point dist | Sample dist |
| Other | 0.788 | 1.000 |
| B | 0.100 | 0.797 |
| A-gamma | 0.057 | 0.916 |
| A-beta | 0.047 | 0.996 |
| A-delta | 0.007 | 0.130 |
Tableau 3. Un tableau de distribution des fréquences et de statistiques mettant en évidence la représentation des différents types de fibres dans les données. La distribution des points indique le rapport entre le nombre de points de données étiquetés comme appartenant à un type de fibre donné et le reste des points. De manière similaire, la distribution des échantillons présente ce rapport au niveau des échantillons, en considérant 129 768 comme le nombre total d’échantillons du jeu de données.
Défis et analyse
Nous avons utilisé ces résultats pour identifier des défis liés aux données et à la modélisation, ce qui nous permet de définir des stratégies pour l'acquisition future de données. Dans les jeux de données d'entraînement, nous avons observé :
- une variabilité inter-sujets imperceptible mais présente,
- un manque de représentativité pour certains types de fibres nerveuses, et
- une redondance d’échantillons similaires au sein de chaque sujet.
L’augmentation de la complexité des modèles ou la simple modification des stratégies d’entraînement ne suffisent pas à surmonter ces limitations.
Pour étayer ces constats, nous avons effectué une analyse par bootstrapping, en augmentant progressivement le nombre de sujets dans les données d’entraînement. Les résultats ont confirmé nos observations et ont mis en évidence l’impact positif de la diversité inter-sujets, bien qu’avec un effet limité. Cela nous a fourni une motivation claire pour collecter davantage de données variées.
Dans ce but, et afin d’intégrer les modèles dans un environnement de collecte de données en temps réel, nous avons développé un outil d’annotation interactif permettant de corriger les prédictions imprécises du modèle. Nous pouvons ainsi utiliser une boucle de rétroaction pour améliorer le modèle en temps réel pendant la session.
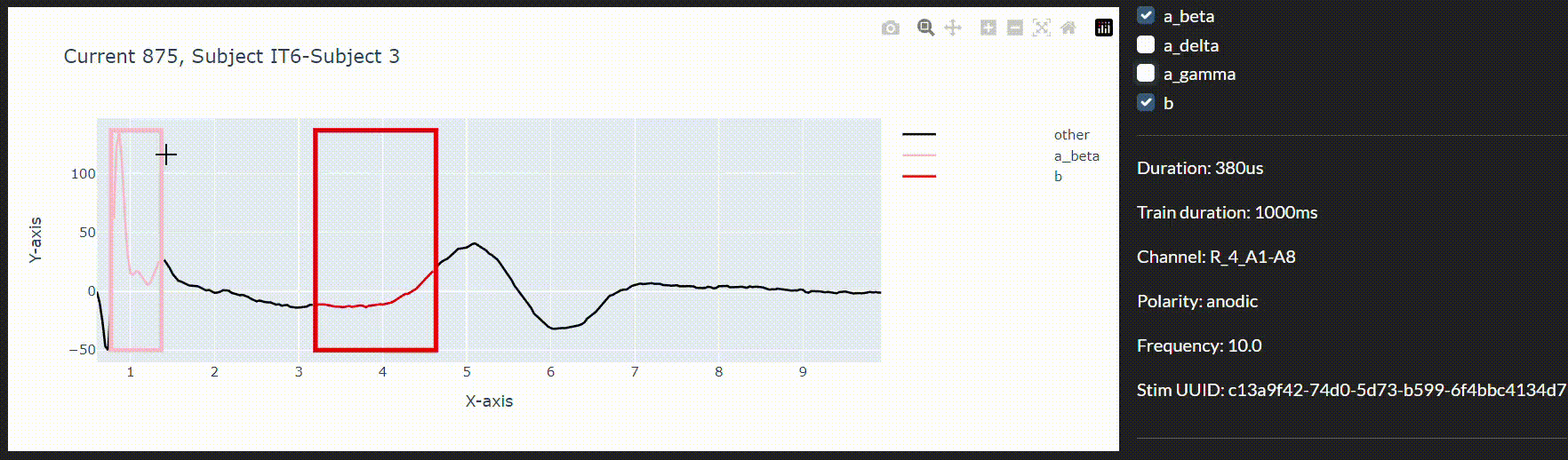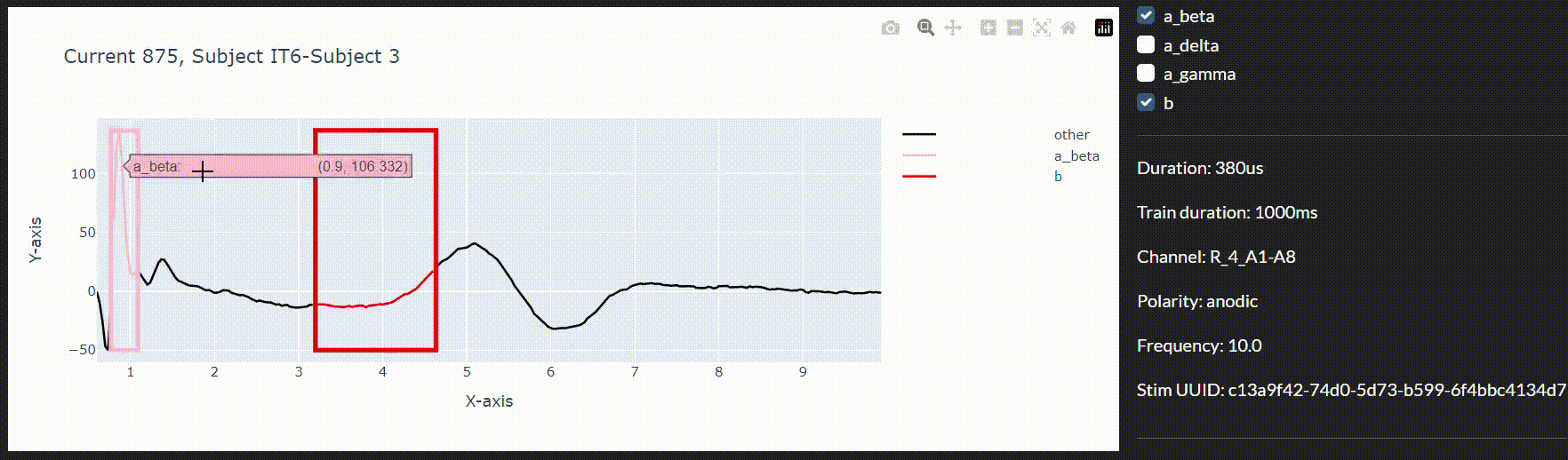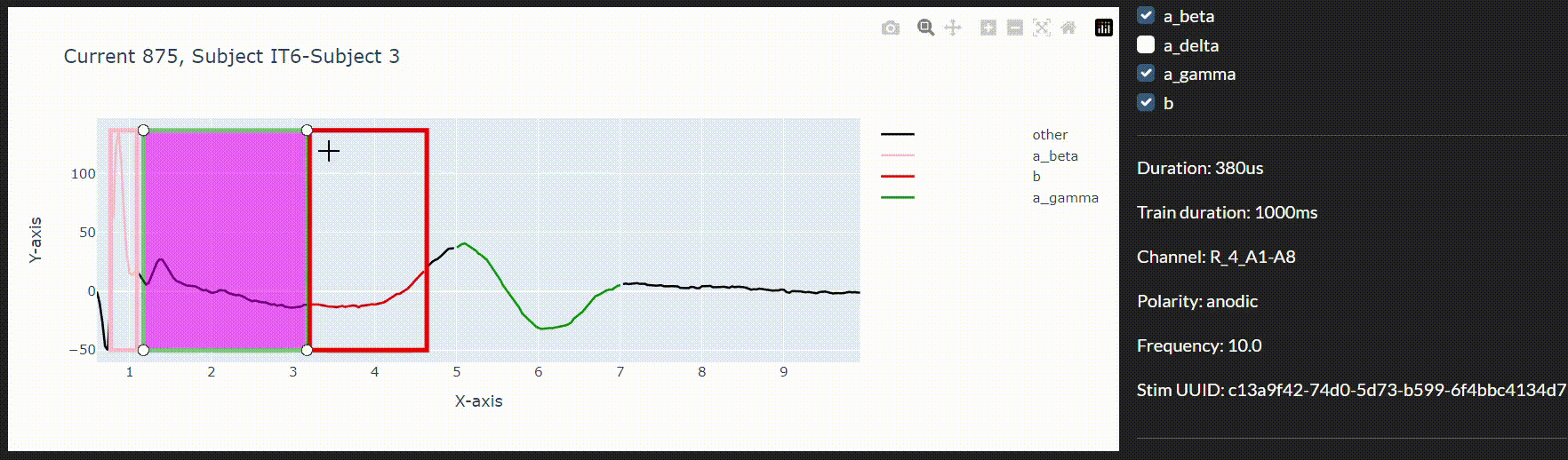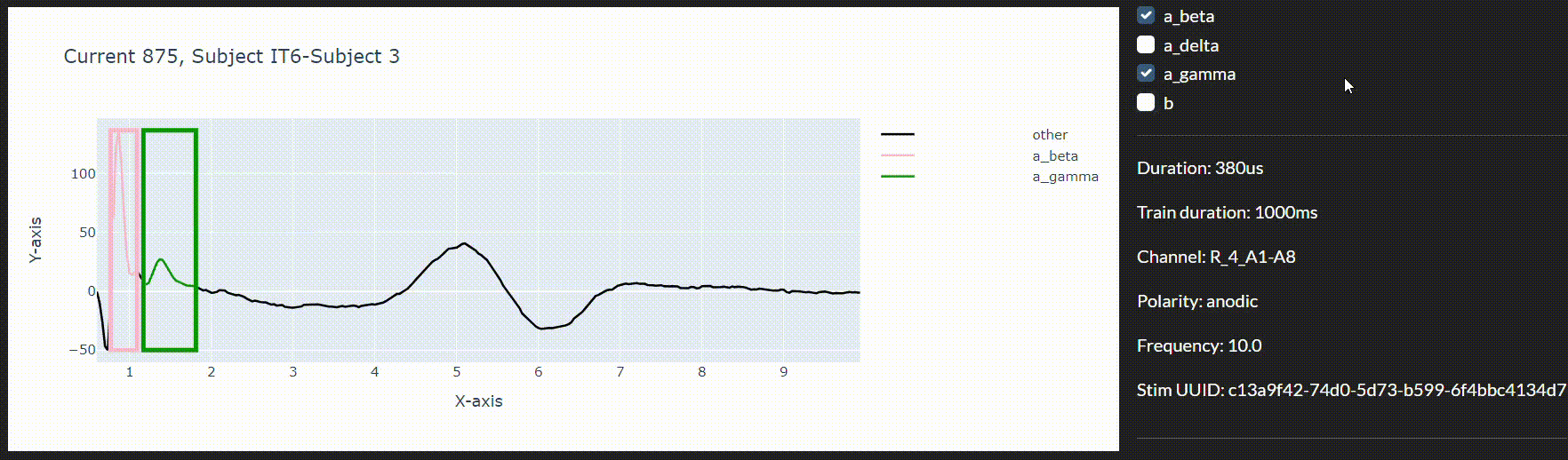
Conclusion
Notre étude sur l’utilisation de l’apprentissage automatique pour automatiser la segmentation des eCAP constitue une preuve de concept réussie démontrant le potentiel de l’acquisition de données à grande échelle pour faciliter le développement de thérapies de stimulation du nerf vague en boucle fermée.
Comparé aux méthodes manuelles, notre modèle offre une adaptabilité plus rapide aux sujets inconnus et une approche plus fiable et fondée sur les données pour répondre à la tâche.
De plus, les analyses complémentaires renforcent la justification opérationnelle et scientifique pour réaliser davantage d’essais et collecter des données plus ciblées et de meilleure qualité.
Enfin, l’outil d’annotation ainsi que le flux de travail proposé pour le réentraînement en temps réel accélèrent considérablement nos efforts dans cette direction.
Références
[1] Berthon, A., Wernisch, L., Stoukidi, M., Thornton, M., Tessier-Lariviere, O., Fortier-Poisson, P., Mamen, J., Pinkney, M., Lee, S., Sarkans, E., Annecchino, L., Appleton, B., Garsed, P., Patterson, B., Gon- shaw, S., Jakopec, M., Shunmugam, S., Edwards, T., Tukiainen, A., Jennings, J., Lajoie, G., Hewage, E., and Armitage, O. Using neural biomarkers to personalize dosing of vagus nerve stimulation. bioRxiv, 2023. doi: 10.1101/2023.08.30.555487. URL https://www.biorxiv.org/content/ early/2023/09/01/2023.08.30.555487.