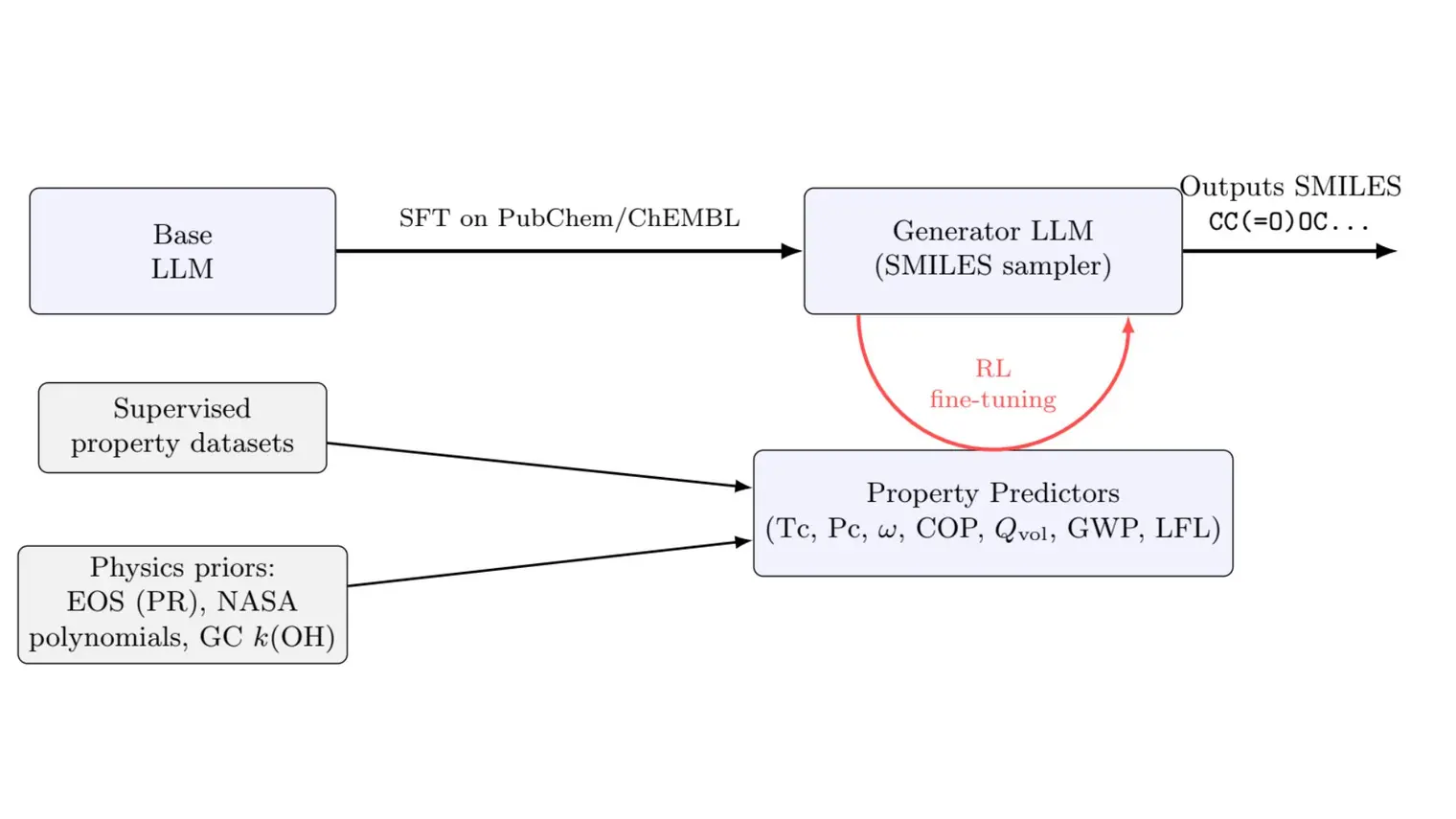
Note de la rédaction : Article fondé sur des textes publiés en tant que communications au NeurIPS 2022 et à l’ICML 2023.
Le problème
La création d’une nouvelle entité dotée de caractéristiques précises occupe une place considérable dans le domaine de la recherche scientifique. Imaginez par exemple que vous conceviez un robot dans le but de le faire courir plus vite.

L’évaluation du rendement de chaque itération du concept peut prendre du temps et s’avérer coûteuse. C’est pourquoi nous nous appuyons souvent sur des ensembles de données existants de différents concepts et sur leurs indicateurs de performance correspondants – dans le cas présent, les tailles et les vitesses des robots. Dans cet article, nous nous penchons sur l’exploitation de cet ensemble de données statiques pour développer un concept optimal, un processus connu sous le nom d’optimisation hors ligne basée sur un modèle.
Travaux connexes et motivation
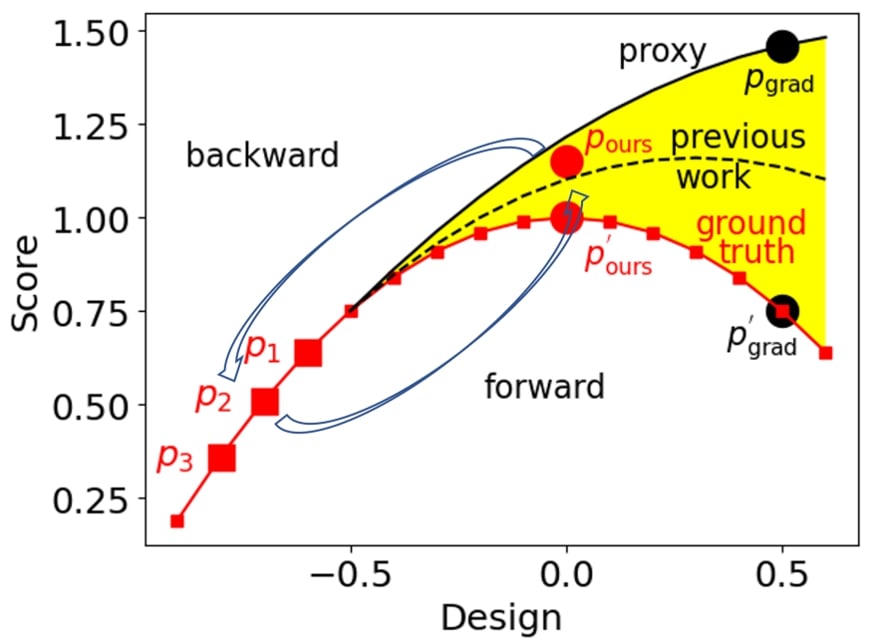
Comme illustré ci-dessus, avec trois paires taille-vitesse de robot comme ensemble de données statiques, une approche courante consiste à entraîner un proxy avec des paramètres pour s’adapter à l’ensemble de données statiques (paires taille-vitesse de robot). Ce proxy entraîné sert d’approximation à la fonction réelle (groundtruth) qui prédit la vitesse du robot en fonction de sa taille.
Par la suite, la taille optimale du robot peut être obtenue en optimisant la taille du robot pour maximiser la fonction proxy selon la direction du gradient. Pourtant, comme nous l’avons vu plus haut, le proxy entraîné sur les trois paires taille-vitesse de robot surestime la fonction réelle, et la taille de robot apparemment optimale est assortie d’une faible vitesse .
Des études antérieures ont tenté de mieux adapter le proxy à la fonction réelle du point de vue du modèle et de rapprocher le proxy de la réalité. La taille optimale de robot peut ensuite être obtenue par ascension du gradient en fonction du nouveau proxy.
Apprentissage bidirectionnel
La méthode que nous étudions dans cet article ne vise pas à entraîner un meilleur proxy plus proche de la réalité, mais plutôt à garantir que la paire taille-vitesse de robot optimale peut servir à prédire les paires taille-vitesse de robot disponibles . Comme illustré ci-dessus, en veillant à ce que la paire taille-vitesse de robot optimale puisse prédire les paires taille-vitesse de robot disponibles (mapping inverse) et vice versa (mapping direct), la paire taille-vitesse de robot optimale distille davantage de renseignements à partir de , ce qui rend plus aligné sur , menant à une vitesse de robot élevée pour . Cette approche permet de rapprocher le design optimal de l’ensemble des données, ce qui se traduit par un concept plus efficace.
Le mapping direct et le mapping inverse permettent d’aligner la paire taille-vitesse de robot optimale sur les paires taille-vitesse de robot disponibles et la perte peut être écrite de manière compacte comme suit : où nous cherchons à optimiser la taille de robot . Bien que cet exemple ne concerne que le concept du robot, il peut également s’appliquer à d’autres domaines.
Résultats expérimentaux
En termes simples, nos objectifs expérimentaux consistent à créer et à améliorer différents concepts, dans le but ultime de maximiser leur efficacité ou leur rendement. Voici une liste des objectifs spécifiques de chaque expérience :
- Pour les robots de type Fourmi et de type D’Kitty, nous tentons de déterminer la morphologie (c.-à-d. la forme ou structure corporelle) qui permettra à ces robots de se déplacer le plus rapidement possible.
- Lors de l’expérience sur les supraconducteurs, nous concevons des matériaux dans le but d’atteindre la température critique la plus élevée possible, à savoir la température en dessous de laquelle le matériau devient supraconducteur.
- Dans l’expérience Hopper Controller, nous ajustons le « cerveau » (ou la politique du réseau de neurones) d’un robot de type sauteur, qui dicte ses actions, dans le but d’améliorer son rendement global.
- Pour l’expérience GFP, nous examinons différentes séquences de protéines afin de trouver celle ayant la plus grande intensité de fluorescence, qui mesure l’efficacité de la protéine.
- Dans l’expérience TFBind8, notre objectif consiste à découvrir les séquences d’ADN qui affichent les résultats d’activité de liaison les plus élevés, ce qui signifie qu’elles sont les plus aptes à s’attacher à des cibles précises.
- Enfin, dans l’expérience UTR, nous essayons d’identifier les séquences d’ADN qui produisent les niveaux d’expression les plus élevés, ce qui nous indique le degré d’activité d’un gène particulier.
En substance, nous essayons de trouver les concepts ou les séquences les plus efficaces dans chacune des sept tâches (deux en robotique, et une pour chaque objectif spécifique) afin d’atteindre le potentiel maximal du sujet traité, qu’il s’agisse d’un robot, d’une protéine, d’une séquence d’ADN ou d’un supraconducteur. Afin d’évaluer le succès de nos expériences, nous rapportons le résultat normalisé selon la réalité terrain (groundtruth-normalized) le plus prometteur, et ce, pour chaque tâche. Ce score nous fournira une mesure standardisée du rendement de chaque concept par rapport aux autres, ce qui nous permettra de disposer d’un indicateur comparatif clair.
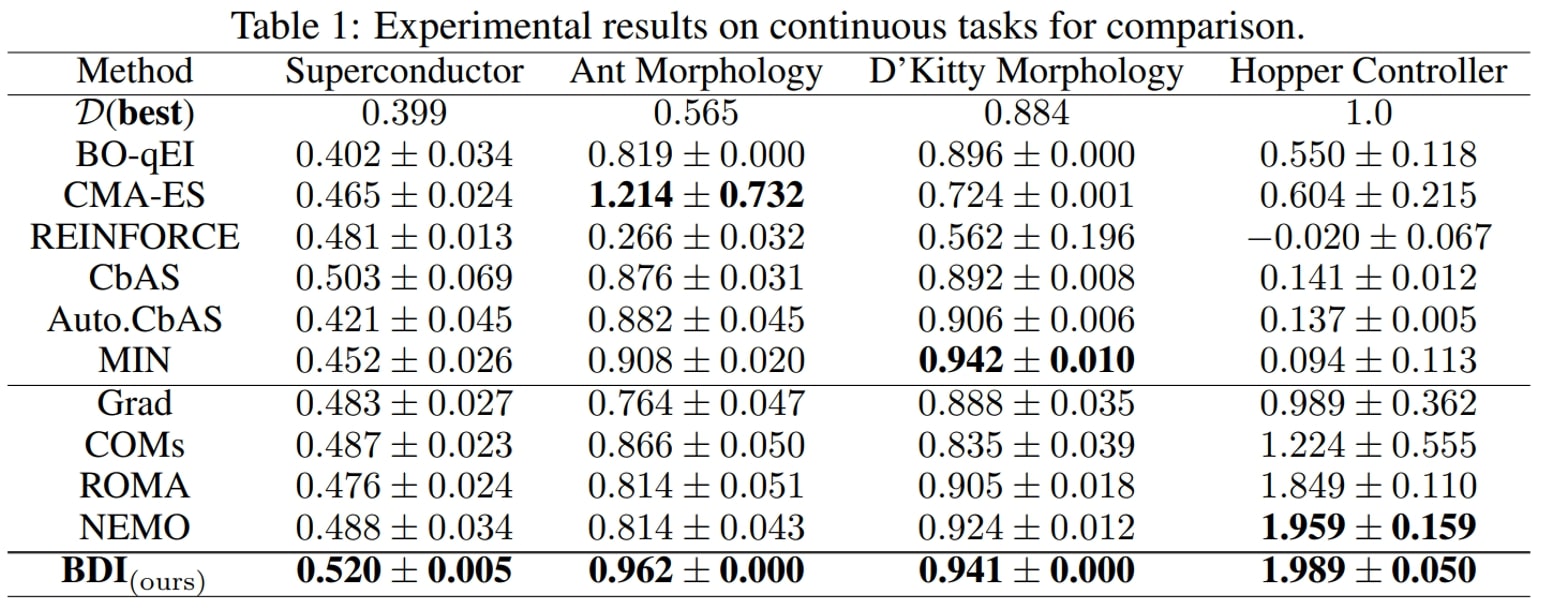
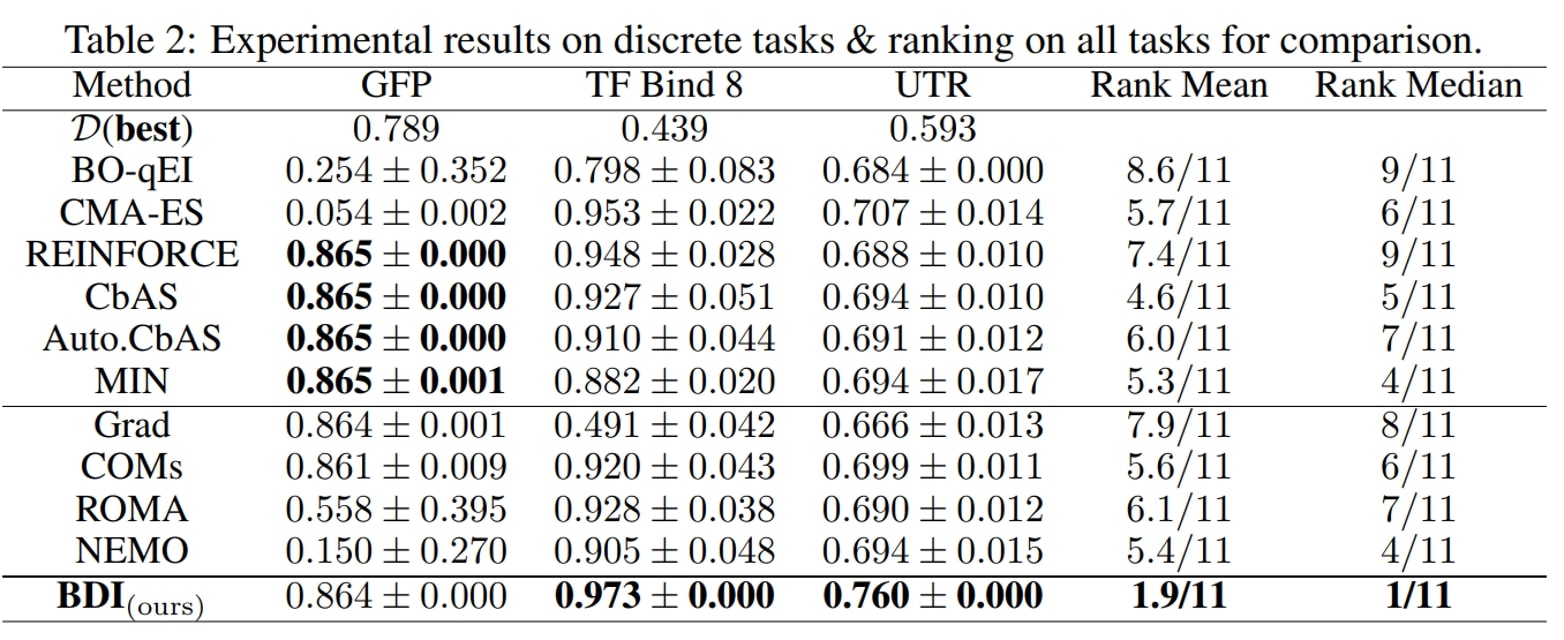
Nous formulons les principales observations suivantes :
- Tout d’abord, le modèle BDI obtient les meilleurs classements, affichant un classement moyen de 1,9 et un classement médian de 1 parmi les 11 méthodes, et obtient le meilleur rendement pour six des sept tâches.
- De plus, par rapport à la méthode naïve Grad, BDI permet de réaliser des gains constants pour chacune des quatre tâches. En particulier pour la tâche TFBind8, la méthode BDI est bien meilleure que la méthode Grad : son rendement est supérieur de 94,3 %. Cela suggère que BDI peut aligner le concept ayant obtenu le score le plus élevé avec l’ensemble de données statique et, ainsi, atténuer le problème des données hors distribution.
- En outre, en ce qui concerne COM, ROMA et NEMO, qui imposent tous un préalable au modèle DNN, ils obtiennent généralement un meilleur rendement que la méthode Grad, mais un rendement inférieur à celui de BDI.
- Enfin, les méthodes fondées sur les modèles génératifs CbAS (0,141), Auto.CbAS (0,137) et MIN (0,094) ne parviennent pas à réaliser des tâches sur des données hautement dimensionnelles comme Hopper (D=5126), car les distributions de telles données sont plus difficiles à modéliser. Par rapport aux méthodes fondées sur un modèle génératif, BDI permet d’obtenir de meilleurs résultats tout en étant beaucoup plus simple.
Du concept général à la séquence biologique
Nous avons également intégré des modèles de langage préentraînés avec BDI afin d’améliorer le rendement des tâches impliquant des séquences biologiques. La nouvelle méthode, connue sous le nom de BIB, a été mise à l’essai dans le cadre de diverses tâches liées aux séquences d’ADN et de protéines.
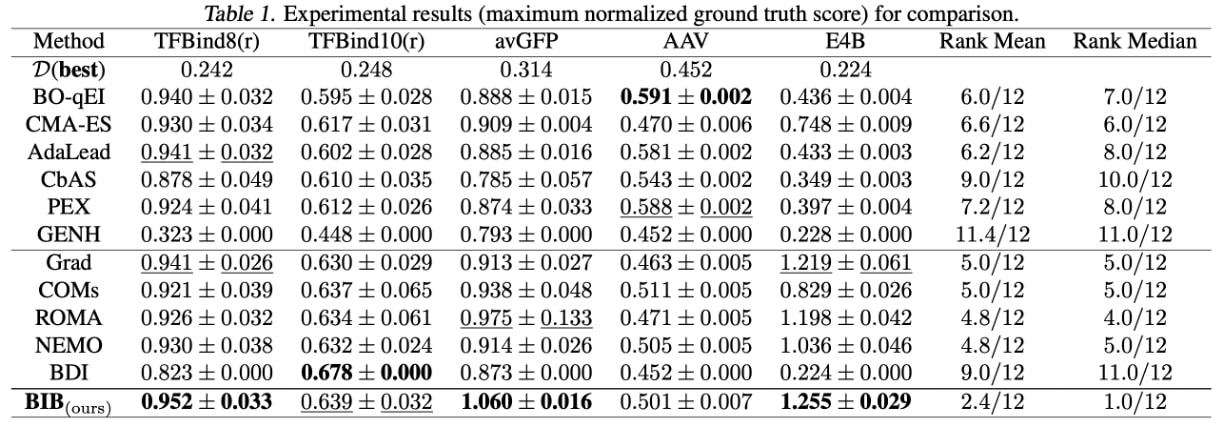
Nous formulons les observations suivantes :
- Tout d’abord, BIB obtient le meilleur rendement pour trois des cinq tâches considérées et affiche les meilleurs classements (classement moyen de 2,4 sur 12 et classement médian de 1,0 sur 12).
- En outre, BIB surpasse systématiquement la méthode Grad pour toutes les tâches : 0,952 > 0,941 dans TFBind8(r), 0,639 > 0,630 dans TFBind10(r), 1,060 > 0,873 dans avGFP, 0,501 > 0,452 dans AAV et 1,255 > 0,224 dans E4B. Cela prouve que BIB peut atténuer efficacement le problème de données hors distribution.
- Par ailleurs, BIB surpasse BDI dans quatre des cinq tâches considérées : 0,952 > 0,823 dans TFBind8(r), 0,639 < 0,678 dans TFBind10(r), 1,060 > 0,913 dans avGFP, 0,501 > 0,463 dans AAV et 1,255 > 1,219 dans E4B. Cela démontre l’efficacité du modèle de langage biologique préentraîné.
- Enfin, les méthodes fondées sur le gradient sont moins efficaces pour la tâche AAV. L’une des raisons possibles est que l’espace de conception d’AAV () est beaucoup plus petit que ceux d’avGFP () et d’E4B (), ce qui rend la modélisation générative et les algorithmes évolutionnaires plus appropriés.
Conclusion
BDI présente une solution nouvelle et efficace aux problèmes liés à l’optimisation hors ligne basée sur un modèle. Cette méthode démontre la puissance de l’apprentissage bidirectionnel et résout efficacement certaines limites des techniques précédentes. De plus, l’intégration de modèles de langage préentraînés dans BDI améliore l’optimisation des séquences biologiques, ce qui démontre le potentiel de ces modèles.